根据《自然通讯》发表的研究结果,西北医学的一项研究详细介绍了机器学习模型的开发,该模型可通过碎片模式预测无细胞DNA中的DNA甲基化状态。
DNA甲基化是将甲基添加到DNA分子的生物过程,充当某些基因的“关闭开关”,并且在癌症等疾病中通常会出现功能障碍。
游离DNA(各种细胞过程中残留的少量DNA)可以通过全基因组亚硫酸氢盐测序(当前的黄金标准)进行测量,但这种不完善的过程可能会损坏正在测序的DNA,从而限制了科学家对其进行研究的能力。
“无细胞DNA是这些短的DNA片段:当细胞死亡时,它会将DNA释放到血液中,”生物化学和分子遗传学助理教授YapingLiu博士说。-该研究的通讯作者。“这种位于细胞外的游离DNA代表了细胞死亡特征。”
与正常DNA不同,游离DNA以特定模式断裂,并且与表观遗传状态高度相关,这让刘想知道他是否可以利用游离DNA断裂模式来预测DNA甲基化水平,他说。
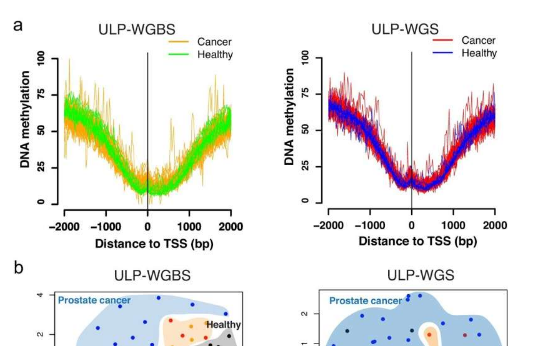
在这项研究中,Liu和他的合作者训练了一个无监督的机器学习模型,利用循环游离DNA片段的特征来分析DNA的小片段(称为CpG位点)。
然后,研究人员使用该模型分析健康患者和不同类型癌症患者的人体血液样本,并对样本进行单独的全基因组测序,以比较模型的准确性。
研究表明,与传统测序相比,该模型能够准确预测DNA甲基化状态,主要位于基因组上CpG丰富的区域。
“临床医生已经通过今天可用的测试生成了大量的无细胞DNA基因组测序数据,”Liu说。“通过我们的模型,我们可以利用这些数据做更多事情,预测DNA甲基化和基因中发生的变化。”
Liu说,该模型还可以准确预测游离DNA来自哪些组织,从而查明各种癌症中出现的异常甲基化特征的起源。
他说,展望未来,刘的实验室将继续开发计算方法,以更好地理解来自游离DNA片段的基因调控信息。
“我们的目标是利用隐藏在游离DNA中的表观遗传信息来了解人类基因组的非编码区域,”刘说,他也是西北大学罗伯特·H·卢里综合癌症中心的成员。“我们不仅希望更早地发现疾病,而且希望有机会了解基因组在那个时间点发生的情况。”
